本文中介绍的是如何在sklearn库中使用PCA方法,以及理解PCA方法中的几个重要参数的含义,通过一个案例来加深理解。

PCA
什么是PCA
主成分分析(Principal components analysis,简称PCA)的思想:
-
将n维特征映射到k维上(k<n),k维是全新的正交特征(新的坐标系)。
-
k维特征称为主元,是重新构造出来的k维特征,而不是简单地从n维特征中去除其余n-k维特征。
-
思想的方法就是降维,用低维的数据去代表高维的数据,也就是用少数几个变量代替原有的数目庞大的变量.
-
把重复的信息合并起来,既可以降低现有变量的维度,又不会丢失重要信息的思想。
Sklearn库中PCA
解释sklearn库中PCA方法的参数、属性和方法。

参数说明
1 | sklearn.decomposition.PCA(n_components=None, copy=True, whiten=False) |
n_components
int, float, None or str
代表返回的主成分的个数,即数据降到几维
- n_components=2 代表返回前2个主成分
- 0 < n_components < 1 代表满足最低的主成分方差累计贡献率
- n_components=0.98,指返回满足主成分方差累计贡献率达到98%的主成分
- n_components=None,返回所有主成分
- n_components=‘mle’,将自动选取主成分个数n,使得满足所要求的方差百分比
copy
bool类型, False/True 默认是True
在运行的过程中,是否将原数据复制。降维过程中,数据会变动。copy主要影响:调用显示降维后的数据的方法不同。
- copy=True时,直接 fit_transform(X),就能够显示出降维后的数据
- copy=False时,需要 fit(X).transform(X) ,才能够显示出降维后的数据
whiten
bool类型,False/True 默认是False
白化是一种重要的预处理过程,其目的是降低输入数据的冗余性,使得经过白化处理的输入数据具有如下性质:
-
特征之间相关性较低
-
所有特征具有相同的方差
svd_solver
str类型,str {‘auto’, ‘full’, ‘arpack’, ‘randomized’}
意义:定奇异值分解 SVD 的方法
- auto:自动选择
- full:传统意义上的SVD
- arpack:直接使用scipy库的sparse SVD实现
- randomized:适用于数据量大,维度多,且主成分比例低的PCA降维
属性atttibutes
-
components_:返回最大方差的主成分。
-
explained_variance_:它代表降维后的各主成分的方差值。方差值越大,则说明越是重要的主成分。
-
explained_variance_ratio_:它代表降维后的各主成分的方差值占总方差值的比例,比例越大,则越是重要的主成分。(主成分方差贡献率)
-
singular_values_:返回所被选主成分的奇异值。实现降维的过程中,有两个方法:
- 特征值分解(需要是方阵,限制多,计算量大)
- 奇异值分解(任意矩阵,计算量小,PCA默认)
-
mean_:每个特征的经验平均值,由训练集估计。
-
n_features_:训练数据中的特征数。
-
n_samples_:训练数据中的样本数量。
-
noise_variance_:噪声协方差
方法Methods
fit(self,X,Y=None)
模型训练,PCA是无监督学习,没有标签,所以Y是None
fit_transform(self,X,Y=None)
将模型和X进行训练,并对X进行降维处理,返回的是降维后的数据
get_covariance(self)
获得协方差数据
get_params(self,deep=True)
返回的是模型的参数
inverse_transform(self,X)
将降维后的数据转成原始数据,不一定完全相同
transform(X)
将数据X转成降维后的数据。当模型训练好后,对于新输入的数据,可以直接用transform方法来降维。
案例分析
1 | import numpy as np |
导入数据作图
学习如何利用sklearn自带的数据
1 | # 生成数据集 |
1 | X |
array([[ 2.38526096, 2.1109917 , 2.23765695],
[ 0.05761939, -0.0117989 , -0.03393958],
[ 3.08207073, 3.19904227, 3.08774759],
...,
[ 3.13804869, 2.86955308, 2.86443838],
[ 2.95413001, 3.42508432, 3.28296407],
[ 2.79195132, 2.94196066, 2.71457256]])
1 | y |
array([3, 1, 0, ..., 0, 0, 0])
1 | fig = plt.figure() |

使用的数据有4个簇
查看方差分布(不降维)
不降维,只对数据进行投影,保留3个属性
1 | from sklearn.decomposition import PCA |
PCA(copy=True, iterated_power='auto', n_components=3, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)
2个属性
查看两个重要的属性:
- 各个主成分的方差占比
- 主成分的方差
1 | print(pca.explained_variance_ratio_) # 降维后的各主成分的方差值占总方差值的比例 |
1 | [0.98318212 0.00850037 0.00831751] # 第一个特征占据绝大多数 |
结论:特征1占据绝大多数
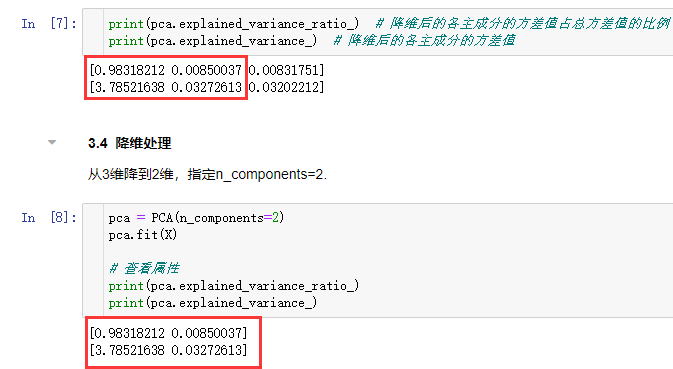
降维处理
从3维降到2维,指定n_components=2.
1 | pca = PCA(n_components=2) # 降到2维 |
[0.98318212 0.00850037]
[3.78521638 0.03272613]
1 | # 查看转化后的数据分布 |

将数据投影到平面上仍然是4个簇
指定主成分占比
比如,想看主成分的占比在99%以上的特征
1 | pca = PCA(n_components=0.99) # 指定阈值占比 |
1 | PCA(copy=True, iterated_power='auto', n_components=0.99, random_state=None, |
1 | print(pca.explained_variance_ratio_) |
1 | [0.98318212 0.00850037] |
第1个特征 和第2个特征之和为:0.9831+0.0085已经超过99%
MLE算法自行选择降维维度
1 | pca = PCA(n_components='mle') |
1 | PCA(copy=True, iterated_power='auto', n_components='mle', random_state=None, |
1 | print(pca.explained_variance_ratio_) |
1 | [0.98318212] |
第一个特征的占比已经高达98.31%,所以算法只保留了第1个特征

